传统手段依赖亚克隆产生转基因外源质粒[1,2],对于生成复杂的DNA构建体具有一定的局限性,其需要使用不同的II型限制性内切酶切割和多次亚克隆连接,效率低且耗时长。为此,催生出了一系列克隆工具如Gateway[3]、In-Fusion[4]和Gibson[5]等,虽然这些方法大大改进了克隆效率,但由于其自身的特点,很难形成一套系统的方法运用于转基因外源质粒的构建,如引入的重组位点可能影响基因和蛋白功能;多轮PCR容易引入突变且这些突变只有在整个连接完成后才能检测出来;线性化片段不利于长期保存和二次利用;难以连接同源性高的相似基因片段等。
鉴于此,2022年12月24日,发育生物学与再生医学研究中心陈静影副研究员与杨昀副教授课题组合作在Journal of Molecular Cell Biology上在线发表了题为“GoldenFish: a rapid and efficient system to customize constructs for zebrafish transgenesis”的研究论文。该研究以斑马鱼为例,创建了一套方便快捷的转基因外源质粒构建系统——GoldenFish,以期快速高效地服务于转基因斑马鱼的构建。

GoldenFish系统以Golden gate克隆技术为原理[6],利用IIS型限制性内切酶识别位点和切割位点位置不同的特点,通过人为设计切割位点,实现黏性末端的自由定制。经过IIS型限制性内切酶消化后,识别位点将从正确连接的DNA构建体中移除,随后,多个DNA片段可以通过悬垂互补进行连接,并按照正确顺序和方向串联起来。
作者首先将外源质粒的组成元件根据不同功能划分为启动子元件库(P library)、目的基因元件库(G library)和终止子元件库(T library)三部分。在IIS型限制性内切酶BsmBI的作用下使每种元件库中的模块带上不同的接头,以方便将各种组件按照对应的接头进行拼接,通过各种排列组合使得转基因质粒像拼装“乐高积木”一样组装为一体(图1),每一个“乐高积木”都可以以质粒形式长期保存及再次使用。

图1 GoldenFish系统中P-G-T构建体的生成
除此之外,GoldenFish技术还可以实现一步构建多转基因外源质粒,即将P-G-T构建体串连起来,在同一条DNA链中由多个启动子来驱动单个或多个基因,生成多转基因构建体(P-G-T)n。多转基因质粒的构建主要分为两步,第一步利用BsmBI和T4连接酶,定向组装各个元件库中的模块,生成1级连接产物。第二步,利用另一种IIS型限制性内切酶BsaI和T4连接酶反应,再将多个1级连接产物进行拼接生成2级连接产物(图2)。目前该系统中的元件库模块已经扩充并涵盖斑马鱼多种组织特异性启动子以及大多数经典荧光蛋白基因,方便后续直接组装连接。
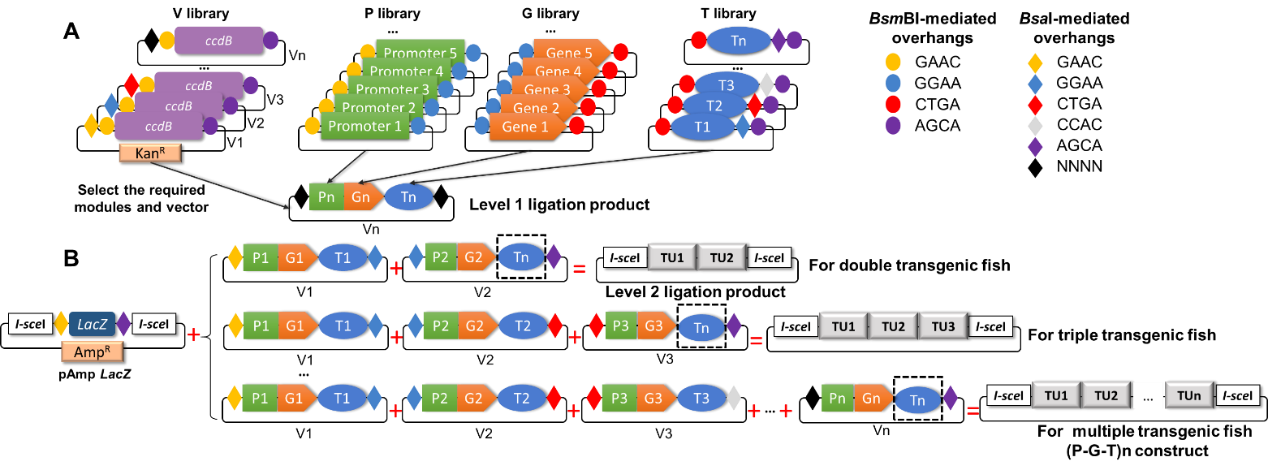
图2 GoldenFish系统中(P-G-T)n结构的构建
该系统具有以下优点:1、能够实现几乎所有类型的外源转基因质粒的构建,包括单转基因,一转多基因,多转基因等,尤其是它可以一步生成包含多个转录单元的构建体。2、“乐高积木”式模块使得DNA构建体可以随时个性化定制,一步完成克隆,显著提高生成转基因构建体的速度和效率。3、存储在同一元件库中的模块可以等效替换和重复使用,为构建不同的DNA质粒提供便利。4、GoldenFish系统除了可以构建斑马鱼转基因系之外,还可以通过合理的改造,将该系统扩展到其他转基因动物。
西南大学博士研究生蒋展眉为论文的第一作者,陈静影副研究员和杨昀副教授为论文通讯作者。
参考文献:
1. Cohen, S.N., et al., Construction of Biologically Functional Bacterial Plasmids in-Vitro. Proceedings of the National Academy of Sciences of the United States of Ameri ca, 1973. 70(11): p. 3240-3244.
2. Roberts, R.J., How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A, 2005. 102(17): p. 5905-8.
3. Hartley, J.L., G.F. Temple, and M.A. Brasch, DNA cloning using in vitro site-specific recombination. Genome Res, 2000. 10(11): p. 1788-95.
4. Sleight, S.C., et al., In-Fusion BioBrick assembly and re-engineering. Nucleic Acids Res, 2010. 38(8): p. 2624-36.
5. Gibson, D.G., et al., Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods, 2009. 6(5): p. 343-5.
6. Engler, C., R. Kandzia, and S. Marillonnet, A one pot, one step, precision cloning method with high throughput capability. PLoS One, 2008. 3(11): p. e3647.